 |
|
|
Архитектура Астрономия Аудит Биология Ботаника Бухгалтерский учёт Войное дело Генетика География Геология Дизайн Искусство История Кино Кулинария Культура Литература Математика Медицина Металлургия Мифология Музыка Психология Религия Спорт Строительство Техника Транспорт Туризм Усадьба Физика Фотография Химия Экология Электричество Электроника Энергетика |
Свойства и функции ДНК-топоизмераз
Рис. 2.25. Механизмы стадий расщепления и лигирования нити ДНК ДНК-топоизомеразами
Рис. 2.26. Доменная структура ДНК-топоизомераз типа IA. А – ДНК-топоизомераза I E. coli, B – ДНК-топоизомераза III E. coli, C – ДНК-топоизомераза IIIa человека. 1 – домен расщепления и прохождения нитей, 2 – домен связывания Zn2+, 3 – С-концевой домен
Рис. 2.27. Трехмерная структура фрагмента остатков 32-509 ДНК-топоизомеразы I E. coli. Указано положение доменов I (остатки 32-63 и 72-157), II (остатки 214-278, 406-433 и 438-475), III (остатки 279-405 и 433-437) и IV (остатки 64-71, 158-213 и 476-560) и междоменных шарниров II/III II/IV, а также каталитического остатка тирозина (Y)
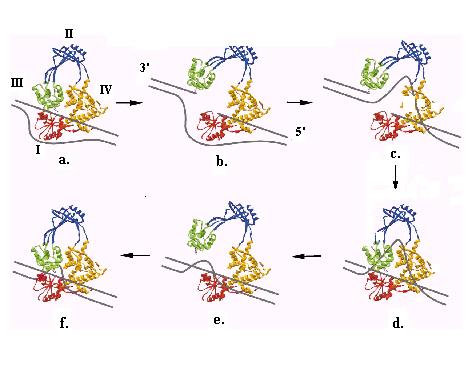
Рис. 2.28. Последовательныке стадии релаксации одного витка негативно суперспирализованной ДНК ДНК-топозомеразой I E. coli. I, II, III и IV – домены топоизомеразы I, указанные на рис. 2.27. Отмечено положение 3’- и 5’-концов разрезаемой нити ДНК
Рис. 2.29. Организация доменов ДНК-топоизомераз типа II. А – ДНК-топоизомераза II S. cerevisiae, B – ДНК-топоизомераза IV E. coli, С – ДНК-гираза E. coli. I – АТФазный домен, II – домен связывания и расщепления ДНК, состоящий из субдоменов A’ и B’, которые соответствуют N-концевой области GyrA иС-концевой области GyrB ДНК-гиразы E. coli, IV – уникальная вставка в С-концевой области субъединицы GyrB ДНК-гиразы E. coli
Рис. 2.30. Трехмерная структура субъединиц ДНК-гиразы E. coli. А – димер N-концевой половины (остатки 1-392) субъединицы GyrB, В – димер фрагмента остатков 30-522 субъединицы GyrA
Рис. 2.31. Механизм переноса Т-сегмента ДНК через расщепляемый G-сегмент ДНК-топоизомеразами типа IIA. I – АТФазные домены (N-ворота), II – домены захвата ДНК, III – области B’, IV - области САР, V – C-ворота. G-сегмент ДНК изображен черной прямой, а Т-фрагмент ДНК – серой прямой. N-ворота открыты на стадиях а-с и закрыты на стадиях d-f, а С-ворота открыты только на стадии f ЛИТЕРАТУРА
1. Matson S.W. DNA helicases of Escherichia coli // Progr. Nucleic Acids Res. Mol. Biol., v. 40, 289-326, 1991 2. Lohman T.M. Helicase-catalyzed DNA unwinding // J. Biol. Chem., v. 268, 2269-2272, 1993 3. West S.C. DNA helicases: new breeds of translocating motors and molecular pumps // Cell, v. 86, 177-180, 1996 4. Lohman T.M., Bjornson K.P. Mechanism of helicase-catalyzed DNA unwinding // Ann. Rev. Biochem., v. 65, 169-214, 1996 5. Patel S.S., Picha K.M. Structure and function of hexameric helicases // Ann. Rev. Biochem., v. 69, 651-697, 2000 6. Hall M.C., Matson S.W. Helicase motifs: the engine that powers DNA unwinding // Mol. Microbiol., v. 34, 867-877, 1999 7. Kersey S.E., Labib K. MCM proteins: evolution, properties, and role in DNA replication // Biochim. Biophys. Acta, v. 1398, 113-136, 1998 8. Tye B.K., Sawyers S. The hexameric eukaryotic MCM helicase: building symmetry from nonidentical parts // J. Biol. Chem., v. 275, 34833-34836, 2000 9. Labib K., Diffley J.F.X. Is the MCM2-7 complex the eukaryotic DNA replication fork helicase? // Curr. Opin. Genet/ Devel., v. 10, 64-70, 2001 10. von Hippel P.H., Delagoutte E. A general model for nucleic acids helicase and their “coupling” within macromolecular machines // Cell, v. 104, 177-190, 2001 11. Chase J.W., Williams K.R. Single-stranded DNA binding proteins required for DNA replication // Ann. Rev. Biochem., v. 55, 103-136, 1986. 12. Lohman T.M, Bujalowski W., Overman L.B. E. coli single strand binding protein: a new look at helix-stabilizing proteins // Trends Biochem. Sci., v. 13, 250-255, 1988. 13. Wold M.S. Replication protein A: a heterodimeric, single stranded DNA-binding protein required for eucaryotic DNA metabolism // Ann. Rev. Biochem., v. 66, 61-92, 1997. 14. Raghunathan S., Ricard C.S., Lohman T.M., Waksman G. Crystal structure of the homo-tetrameric DNA binding domain of Escherichia coli single-stranded DMA binding protein determined by multiwavelength x-ray diffraction on the selenomethionine protein at 2.9-A resolution // Proc. Nat. Acad. Sci. USA, v. 94, 6652-6657, 1997. 15. Shamoo Y., Friedman A.M., Parsons M.R., Konigsberg W.H., Steitz T.A. Crystal structure of a replication fork single-stranded DNA binding protein (T4 gp32) complexed to DNA // Nature, v. 376, 362-366, 1995. 16. Bochkareva E., Belegu V., Korolev S., Bochkarev A. Structure of the major single stranded DNA-binding domain of replication protein A suggests a dynamic mechanism for DNA binding // EMBO Journal, v. 20, 612-618, 2001. 17. Griep M.A. Primase structure and function // Indian J. Biochem. Biophys., v. 32, 171-178, 1995. 18. Arezi B., Kuchta R.D. Eucaryotic DNA primase// Trens Biochem. Sci., v. 25, 572-576, 2000. 19. Frick D.N., Richardson C.C. DNA primases // Ann. Rev. Biochem., v. 70, 39-80, 2001. 20. Aravind L., Leipe D.D., Koonin E.V. Toprim – a conservative domain in type IA and II topoisomerases, DnaG-type primases, OLD family nucleases and RecR proteins // Nucl. Acids Res., v. 26, 4205-4213, 1998. 21. Lehman I.R. DNA ligase: structure, mechanism, and function // Science, v. 186, 790-797, 1974 22. Lindahl T., Barnes D.E. Mammalian DNA ligases // Ann. Rev. Biochem., v. 61, 285-281, 1992. 23. Doherty A.J., Suh S.W. Structural and mechanistic conservation in DNA ligases // Nucleic Acids Res., v. 28, 4051-4058, 2000 24. Shuman S., Schwer B. RNA capping enzyme and DNA ligase: modular architecture and functional implications // Mol. Microbiol., v. 17, 405-410, 1995. 25. Lee J.Y., Chang C., Song H.K. et al. Crystal structure of NAD+-dependent DNA ligase: modular architecture and functional implications // EMBO Journal, v. 19, 1119-1129, 2000. 26. Postow L., Crisona N.J., Peter B.J., Hardy C.D., Cozzarelli N.R. Topological challenges to DNA replication // Proc. Nat. Acad. Sci. USA, v. 98, 8219-8236, 2001. 27. Wang J.C. DNA topoisomerases // Ann. Rev. Biochem., v. 65, 635-692, 1996 28. Berger J.M. Structure of DNA topoisomerases // Biochim. Biophys. Acta, v. 1400, 3-18, 1998 29. Champoux J.J. DNA topoisomerases: structure, function, and mechanism // Ann. Rev. Biochem., v. 70, 369-413, 2001. 30. Tse-Dinh Y.-C. Bacterial and archeal type I topoisomerases // Biochim. Biophys. Acta, v. 1400, 19-27, 1998. 31. Levine C., Hiasa H., Marians K.J. DNA gyrase and topoisomerase IV: biochemical activities, physiological roles during chromosome repkication, and drug sensitivities // Biochim. Biophys. Acta, v. 1400, 29-43, 1998. 32. Nitiss J.L. Investigating the biological functions of DNA topoisomerases in eukariotic cells // Biochim. Biophys. Acta, v. 1400, 63-81, 1998.
ГЛАВА 3. Инициация репликации хромосомной ДНК
3.1. Инициация репликации хромосомы E. coli Белок-инициатор DnaA
Белок DnaA играет ключевую роль в инициации репликации хромосомы у многих бактерий. Он последовательно выполняет 3 главные функции: 1) узнает область начала репликации oriC, последовательно связываясь с нонамерными повторами в ДНК – блоками DnaA; 2) способствует расплетанию легкоплавких АТ-богатых участков ДНК oriC; 3) вербует на расплетенные участки oriC ДНК-геликазу DnaB. У E. coli белок DnaA имеет длину 467 остатков (рис. 3.1) и состоит из 4 определяемых гомологией доменов (I-IV), каждый из которых в разной степени участвует в инициации репликации на oriC. Наиболее консервативными среди бактериальных белков DnaA являются домены III и IV (остатки 130-350 и 351-367 соответственно). Несколько меньше гомология в N-концевом домене I (остатки 1-56), а домены II (остатки 57-129 у E. coli) белков DnaA у разных бактерий сильно различаются по длине и аминокислотной последовательности и иногда рассматриваются как неконсервативные гибкие линкеры, так как вставки или делеции в большей части домена II не влияют на функции. Все белки DnaA имеют модульную структуру. Первые 86 остатков DnaA образуют домен олигомеризации, состоящий из гептадных повторов гидрофобных остатков. Этот домен участвует в образовании мультимерных комплексов белка DnaA на ДНК oriC. Он может обеспечивать и димеризацию других белков. Так, слияние домена олигомеризации DnaA с N-концевым доменом связывания с ДНК репрессора CI фага l придает химерному белку способность к димеризации. Домен олигомеризации DnaA перекрывается с частью домена, связывающего геликазу DnaВ (остатки 24-86). В физическом контакте с DnaВ участвуют также остатки 136-148 белка DnaA. Впрочем, считается, что для эффективной функции погрузки на oriС белка DnaВ требуются почти все области DnaA. Центральный домен III имеет открытую изогнутую структуру ab, типичную для связывающих нуклеотиды белков, и содержит контактный участок для связывания нуклеотидов (остатки 168-236), критическую роль в котором играет остаток лиз178. Белок DnaA может связывать АТФ и АДФ. Связывание АТФ вызывает изменение конформации DnaA. Комплекс DnaA-АТФ абсолютно необходим для инициации репликации и является функционально активным, в отличие от неактивного комплекса с АДФ. Весь домен III обладает характеристической АТФазной активностью, но она низка и требует стимуляции другими белками. Этот домен необходим для локального расплетания ДНК в АТ-богатых областях oriC. Однако гидролиз АТФ не требуется для активности DnaA, и АТФ можно заменить на его негидролизуемый аналог АТФgS. Короткий участок в начале домена IV (остатки 372-381) участвует в регуляторных взаимодействиях DnaA с кислыми фосфолипидами клеточной мембраны. Большая часть домена IV (остатки 374-467) необходима и достаточна для специфического связывания с ДНК. В таком связывании белок DnaA узнает консервативные участки ДНК длиной 9 п.н. – “блоки DnaA” c консенсусной последовательностью 5’-ТТАТ(C/А)САСА. С этими блоками белок DnaA может взаимодействовать кооперативно и образовывать мультимерные комплексы за счет белок-белковых взаимодействий. Домен связывания с ДНК состоит из трех a-спиралей: амфипатических спиралей А и В с основной петлей между ними и длинной спирали С. Мутации во всех этих участках домена связывания с ДНК могут нарушать ассоциацию DnaA с ДНК. Мутации, затрагивающие первые 12 остатков на N-конце спирали С изменяют специфичность узнавания последовательности блока DnaA.
Рис. 3.1. Структура белка DnaA E. coli. I-IV - области гомологии бактериальных белков DnaA; 1 – домен олигомеризации, 2 – области связывания с геликазой DnaВ, 3 – сайт связывания нуклеотидов, 4 – домен АТФазной активности, 5 – участок взаимодействия с мембранными фосфолипидами, 6 – домен связывания с ДНК.
Поиск по сайту: |










 1 56 129 350 429
1 56 129 350 429



 N I II III IV C
N I II III IV C
 1 6
1 6

 2 2 5
2 2 5 3
3 4
4